AI Bootcamp Section1 Review3
Updated:

AI Bootcamp Section1 Review3
PCA
week3을 하면서 내가 아직도 이해가 잘가진 않지만 음… 그러니깐 내용이 머리로는 이해가 가는데 python의 모듈이 돌아가는 과정이 이해가 가진 않지만 그래도 해볼순 있는데 까진 해보려고 한다. 어차피 이건 Review이니깐 PCA는 정확하게 이해하려면 기본적인 학부수준의 선형대수학의 이해가 행되어야 하지만 그것 보다 나는 과정을 Review하고 싶기 때문에 선형대수학의 이해는 건너 띄고 설명 하겠다.
PCA는 단순히 주성분 분석이라기보다는 주성분이 될 수 있는 형태로 내가 가지고 있는 기존 데이터에 어떤 변환을 가하는 것이다.
결론적으로 내가 가지고 있는 데이터에 어떤 기준에 의해서 어떤 변환이 생기게 되고 그 변환으로 인해 ‘주성분’이 추출된다.
그러므로, 이 주성분은 내가 원래 가지고 있는 데이터와 다르다. 변환된 데이터이다.
따라서, 원래 변수가 가지고 있는 의미 즉 열의 의미가 중요한 경우에는 PCA를 사용하면 안 된다.
왜냐하면, 위에서 말했듯이 PCA는 데이터에 변환을 가하는 것이기 때문이다.
- 일단 내가 가장 이해가 가지 않았던 PCA과정의 순서이다.
- Mean centering
- SVD(Singular-value decomposition) 수행
- Scikit-Learn에서 PCA를 계산할 때에도 고유값 분해(eigenvalue-decomposition)가 아닌 특이값 분해(SVD, Singular Value Decomposition)를 이용해 계산한다고 한다. 그 이유는 SVD를 이용하는 것이 계산 속도에 매우 유리하다고 한다.
- PC score 구하기
- PC score를 활용하여 분석 진행
- PC score를 설명변수로 활용하여 분석을 진행합니다.
# 데이터 핸들링을 위한 라이브러리
import pandas as pd
import numpy as np
# 시각화 라이브러리
import matplotlib.pyplot as plt
import seaborn as sns
# Sklearn에 있는 기본 데이터셋을 가져오기 위한 라이브러리
from sklearn import datasets
# sklearn의 PCA를 통해서 쉽게 PCA 적용가능
from sklearn.decomposition import PCA
# 그렇지 않으면 위에서 건너띈 선형대수학의 늪으로 들어가
# eigen_vector, eigen_value를 구해야하는 등 과정이 복잡해진다.
#loading dataset
iris = datasets.load_iris()
df_iris = pd.DataFrame(data= np.c_[iris['data'], iris['target']],
columns= iris['feature_names'] + ['target'])
df_iris.head()
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | target | |
|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | 0.0 |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | 0.0 |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | 0.0 |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | 0.0 |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | 0.0 |
Featrue & Target 확인하기
독립 변수와 종속 변수를 확인한다.
독립 변수가 어느 정도 정규분포를 따르는지, 종속 변수 값들이 적절한지 확인한다.
종속 변수가 99개와 1개 이런 식이면 PCA를 하는 의미가 없다.
독립 변수가 min값과 max값 쪽에 몰려있는 식으로 이분화되어있다면 이 또한 그다지 의미가 없으므로 기본적인 데이터의 상태를 확인한다.
X = iris.data
y = iris.target
feature_names = iris.feature_names
df_x = df_iris.iloc[:, :-1]
df_y = df_iris.iloc[:, [-1]]
sns.pairplot(df_iris, hue="target", height=3)
plt.show();
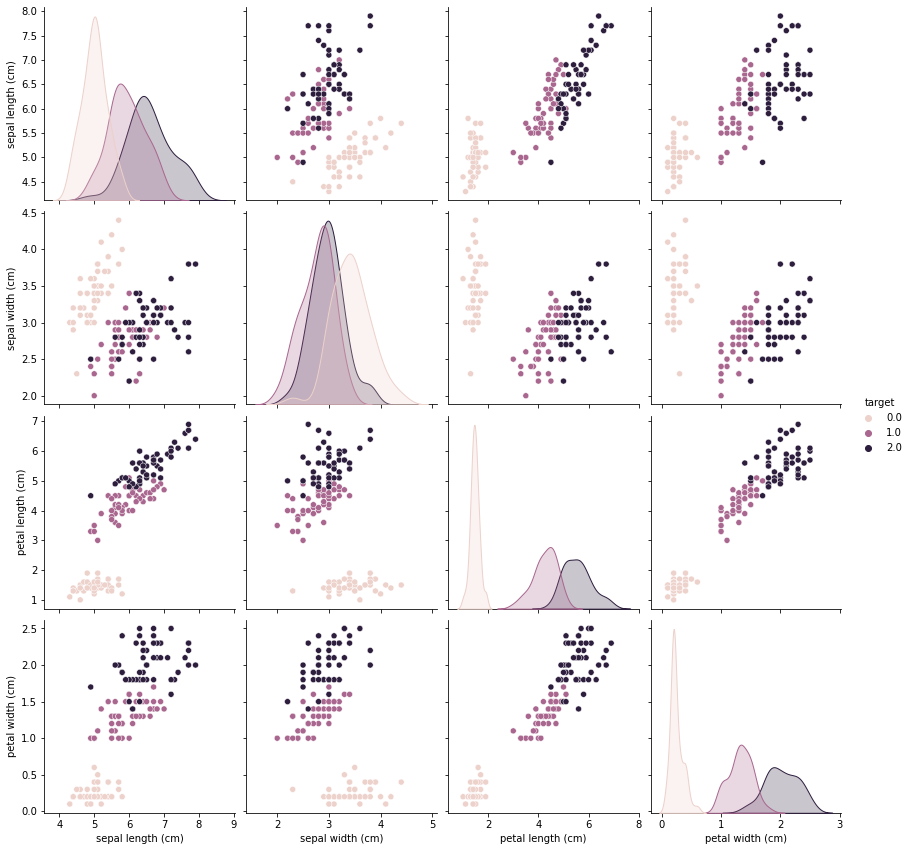
- Target 변수는 0, 1, 2로 범주화되어있다. 또한, 150개가 50개씩 고르게 분포하고 있는 것을 확인할 수 있다.
- Features 들은 정규분포에 가까운 그래프를 그려주는 것을 확인할 수 있다.
PCA( Principal Component Analysis)
# sklearn을 통해서 PCA 객체 생성 및 PC값을 구할 수 있다.
pca = PCA()
pca.fit(X)
PC_score = pca.transform(X)
PC_score[:5]
array([[-2.68412563e+00, 3.19397247e-01, -2.79148276e-02,
-2.26243707e-03],
[-2.71414169e+00, -1.77001225e-01, -2.10464272e-01,
-9.90265503e-02],
[-2.88899057e+00, -1.44949426e-01, 1.79002563e-02,
-1.99683897e-02],
[-2.74534286e+00, -3.18298979e-01, 3.15593736e-02,
7.55758166e-02],
[-2.72871654e+00, 3.26754513e-01, 9.00792406e-02,
6.12585926e-02]])
- Transform을 통해서 아주 쉽게 PC값들을 구할 수 있다.
- array 형식으로 각 성분들의 값이 나타나는 것을 확인할 수 있다.
# pca의 eigen_vector
pca.components_
array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102],
[-0.58202985, 0.59791083, 0.07623608, 0.54583143],
[-0.31548719, 0.3197231 , 0.47983899, -0.75365743]])
- pca를 통해 얻은 eigen_vector를 components_를 통해서 확인할 수 있다.
PC 값의 설명력 정도
pca.explained_variance_
array([4.22824171, 0.24267075, 0.0782095 , 0.02383509])
- PC값이 클수록 설명력이 높다.
- 첫 번째, PC 값이 가장 크므로 가장 설명력이 높은 축일 것으로 생각할 수 있다.
- 마지막 두 개의 PC를 보면 값이 낮다.
- 마지막 값의 경우는 약 170배 정도의 설명력 차이가 나는 것을 알 수 있다.
즉, 거의 설명력이 없다고 생각할 수 있다. $\Rightarrow$ 그렇다고 전혀 없는 것은 아니다.
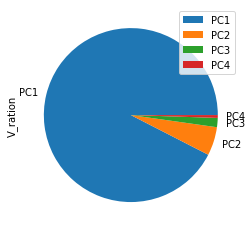
ratio = pca.explained_variance_ratio_
ratio
array([0.92461872, 0.05306648, 0.01710261, 0.00521218])
- PC1 이 92%의 설명력을 가지고 PC4가 0.5%의 설명력을 가지고 있다.
df_v = pd.DataFrame(ratio, index=['PC1','PC2','PC3','PC4'], columns=['V_ration'])
df_v.plot.pie(y='V_ration')
df_v
| V_ration | |
|---|---|
| PC1 | 0.924619 |
| PC2 | 0.053066 |
| PC3 | 0.017103 |
| PC4 | 0.005212 |
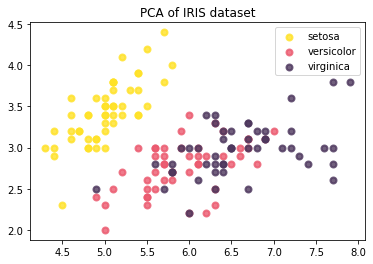
# PCA전 데이터의 scatterplot
plt.figure()
colors = ['#ffe227', '#eb596e', '#4d375d']
lw = 2
for color, i, target_name in zip(colors, [0,1,2] , iris.target_names):
plt.scatter(X[y == i, 0], X[y == i, 1], color=color, alpha=.8, lw=lw,
label=target_name)
plt.legend(loc='best', shadow=False, scatterpoints=1)
plt.title('PCA of IRIS dataset')
Text(0.5, 1.0, 'PCA of IRIS dataset')
# pca후 데이터의 scatterplot
for color, i, target_name in zip(colors, [0,1,2] , iris.target_names):
plt.scatter(PC_score[y == i, 0], PC_score[y == i, 1], color=color, alpha=.8, lw=lw,
label=target_name)
plt.legend(loc='best', shadow=False, scatterpoints=1)
plt.title('PCA of IRIS dataset')
Text(0.5, 1.0, 'PCA of IRIS dataset')
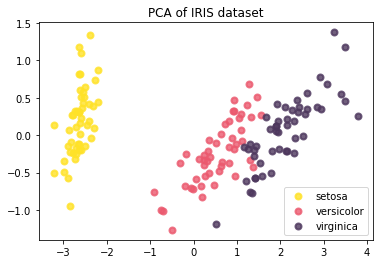
- 확실히 이전 scatter plot 보다 데이터들이 좀 더 구분이 잘 되는 것을 볼 수 있다.


Leave a comment